近日,日本京都大學博士畢業生、目前在美國芝加哥大學從事博士後研究的錢年超和合作者開發出一種全新的空間轉錄組技術——立體 DNA 顯微鏡。

圖 | 錢年超(來源:錢年超)
研究中,他們成功重構了 24hpf 斑馬魚胚胎的三維形態,並以三維方式重現了 Stereo-seq 捕獲的空間基因表達模式,證明該技術在構建組織樣品三維空間轉錄組上的可行性。
另外,他們還開發了一種名爲測地譜嵌入(GSE,Geodesic Spectral Embeddings)的數據降維方法,其能處理測序數據以及重構斑馬魚胚胎的圖像,爲非線性大數據的降維提供了一種新工具。
之所以能夠實現以上成果,是因爲他們在無需依賴微流控系統、光學顯微鏡等特殊設備和先驗知識的前提下,使用滾環擴增這一等溫擴增方法產生的 DNA 納米球可以將 RNA 的遺傳信息進行空間編碼,進而能夠使用 DNA 分子標記 DNA 納米球的鄰近關係,而後可以針對 DNA 進行測序和數據分析。
論文發表之後,錢年超等人受到 Nature Biotechnology 期刊編輯邀請發表了一份研究簡報,該簡報由錢年超等論文作者、期刊編輯和行業專家共同完成的,是對本次成果的概括和評價。在簡報中,專注於單細胞空間蛋白質組學的生物技術公司的瑞典公司 Pixelgen Technologies 的 CEO 西蒙・弗雷德裏克松(Simon Fredriksson)教授評價稱:“這項工作爲三維、無偏見地分析生物學提供了一條途徑,這將是一項偉大的成就。在沒有光和熒光團的情況下定位生物分子具有遠遠超出傳統顯微鏡的潛在可擴展性。”
目前,本次立體 DNA 顯微鏡可以獲得 24hpf 斑馬魚胚胎的三維空間基因表達數據,但是仍然還沒有實現單細胞分辨率。未來,他們將會持續優化這一技術。
(來源:Nature Biotechnology)
若干年後,如能實現單細胞分辨率,可能產生潛在的應用有:
- 首先,目前立體 DNA 顯微鏡僅能獲得三維空間轉錄組信息,但是由於該技術有很好的可擴展性,因此未來有望實現空間基因組、空間蛋白組、空間 ATAC-seq 等三維空間多組學以及三維時空組信息的檢測;
- 其次,本次成果可以提高檢測腫瘤微環境的維度,確定完整組織中與癌細胞相互作用的免疫細胞類型,發現新的腫瘤浸潤免疫細胞;
- 再次,可以解析胚胎髮育過程中細胞之間的相互作用,進而發掘決定細胞命運的關鍵微環境組分;
- 最後,可以與電子顯微鏡互補,繪製大腦的連接組。
(來源:Nature Biotechnology)

開發立體 DNA 顯微鏡,解決現有空間轉錄組技術的侷限性
在開展本次研究之前,錢年超主要研究發育和病毒感染過程中膜蛋白的功能和分子機制。在研究膜蛋白的分子機制過程中,錢年超發現他所研究的三種膜蛋白 TRIC-B、TRPM7 和 TMEM120A 均非獨立發揮作用,而是都要與周圍的其他蛋白質或者小分子相互作用才能完成一定的功能。這讓他意識到在微米甚至納米層面上,膜蛋白周圍環境中的生物分子對其正常功能的重要性。
但是,當時研究單個膜蛋白的分子機制至少需要 2 年,主要原因是當時所用的技術手段太侷限。比如,傳統的免疫熒光染色和熒光原位雜交等技術,雖然可以定位已知蛋白質、RNA、DNA 等生物大分子,並能根據已有文獻和研究者經驗去驗證可能與它們相互作用的其他分子,然而往往通量較低即一次可檢測的分子數量較少,而且只能檢測已知目標。使用單細胞轉錄組技術固然可以高通量地檢測單細胞中的 RNA 分子,但是無法確定它們之間的相互作用。
顧名思義,相互作用——就是在空間上接近並互相影響,所以如能獲得生物分子的空間信息,就可以在一定程度上推定它們之間的相互作用。2016 年,空間轉錄組概念被提出之後,空間轉錄組技術很快就被 Nature Methods 評爲“2020 年年度技術”,至今已經進入快速發展時期。而且隨着空間轉錄組學的發展,腫瘤微環境等方向已經成爲生命科學領域的研究熱點。
空間轉錄組技術之所以能夠迅速崛起和得到廣泛應用,就是因爲該技術既可以高通量地檢測 RNA 和細胞類型,又可以保留它們在組織內的原位空間信息。它可以爲組織樣品繪製一張詳細的“地圖”來展示 RNA 在其中的空間位置,這樣就能推測細胞-細胞之間、RNA-RNA 之間的相互作用。錢年超表示:“之所以用‘推測’,沒有用‘明確’,是因爲目前的空間轉錄組技術還有很多侷限性。”
這些侷限性主要是:
- 首先,由於沒有單細胞分辨率,因此需要使用複雜的算法來進行細胞分割;
- 其次,只能檢測 2D 組織切片,而且由於需要在 2D 平面上標記 RNA 的空間信息,導致現有標記技術都需要把組織強行地像素化。雖然可以把連續 2D 組織切片的空間轉錄組數據整合成 3D 形式,但是在切片製備的過程中 RNA 難免會丟失或錯位,同時數據整合過程也極具挑戰性。另外,即使 2D 組織切片也會有一定的厚度,也就是說它並不是純粹的 2D 平面,所以不同細胞的 RNA 難免會重疊投射到 2D 平面上;
- 再次,需要特殊的儀器設備或試劑,導致應用成本較高。
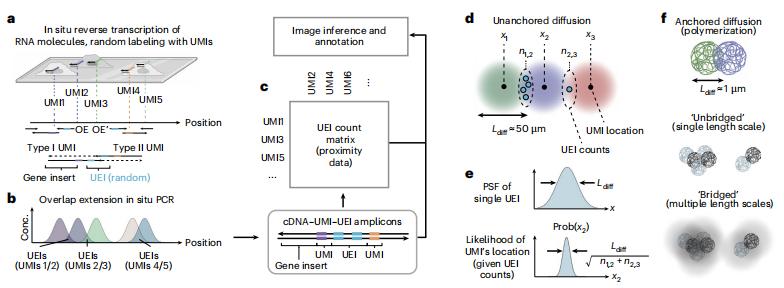
這些侷限性共同限制了該技術的可擴展性和進一步普及。2019 年,美國芝加哥大學教授約書亞·溫斯坦(Joshua A. Weinstein)教授和團隊(即錢年超目前的合作導師)發表了一篇 Cell 論文並提出“DNA 顯微鏡”這一概念。DNA 顯微鏡是將 RNA 的遺傳信息通過聚合酶鏈式反應(PCR,Polymerase Chain Reaction)來原位擴增並用分子識別碼(UMI,Unique Molecular Identifier)標記爲 DNA。
隨着 PCR 反應的進行,DNA 會以 RNA 爲中心向外擴散形成 DNA polonies,然後通過重疊延伸 PCR 將相鄰的 DNA polonies 用事件識別碼(UEI,Universal Event Identifiers)來串聯標記,最後針對所得的串聯 DNA 分子進行測序和數據分析,進而重構出 RNA 的相對空間位置。
當時,錢年超正在清華大學做博士後,看到這篇論文時給他的第一印象就是很酷。反覆閱讀以後,他意識到 DNA 顯微鏡或許能夠克服現有空間轉錄組技術的缺陷。於是,錢年超申請並加入溫斯坦教授團隊,希望能把 DNA 顯微鏡應用到 3D 組織樣品中。但是,當時的 DNA 顯微鏡受到 PCR 條件的限制,因此只能用於 2D 培養細胞中。在和溫斯坦教授討論之後,錢年超決定使用等溫擴增方法來替代 PCR,以便能夠重新設計 DNA 顯微鏡。研究中,他和合作者嘗試了環介導等溫擴增(LAMP,Loop-Mediated Isothermal Amplification)、滾環擴增(RCA,Rolling Circle Amplification)兩種等溫擴增方法,最後發現 RCA 的效果最好。
基於此,錢年超等人通過展開本次研究,旨在開發立體 DNA 顯微鏡來解決現有空間轉錄組技術的侷限性,以便實現真正意義上的具有單細胞分辨率的 3D 空間轉錄組技術,並使用該技術去研究在發育和病毒感染過程中,生物大分子和細胞的空間微環境以及它們的相互作用。
(來源:Nature Biotechnology)

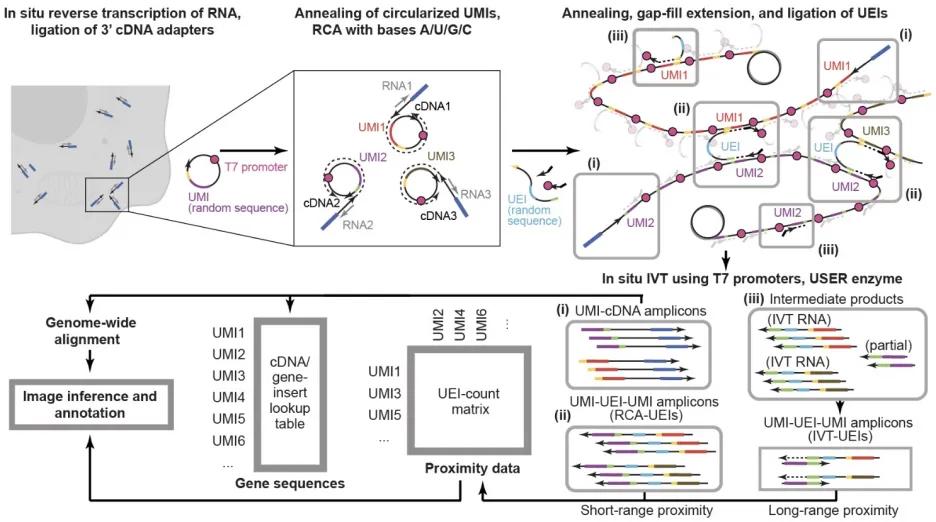
通過 DNA 納米球來標記 RNA 的空間信息
由於溫斯坦教授此前發表的 Cell 論文僅僅使用 DNA 顯微鏡檢測了培養細胞的甘油醛 - 3 - 磷酸脫氫酶和β- 肌動蛋白的空間分佈,並重構了培養細胞的形態。所以,在本次研究的初期,錢年超直接將 DNA 顯微鏡用於三維組織樣品。具體來說,他使用多聚胸苷引物(poly(T),polythymidine)替換了上述 Cell 論文中所使用的基因特定引物來逆轉錄 RNA。然後,他進行了 DNA 顯微鏡的後續操作,藉此成功重構出細胞在培養皿中的分佈,並構建了它的二維空間轉錄組。隨後,他們使用 DNA 顯微鏡檢測了 24hpf 斑馬魚胚胎的空間轉錄組,但是所生成的可用於測序的 DNA 產量很低,完全無法用於重構斑馬魚胚胎的形態。
經過反覆推敲他們意識到,PCR 過高的變性溫度以及快速的升溫過程和降溫過程,都會影響 PCR 反應在三維胚胎中的順利進行,也會影響反應產物在樣品中的均勻擴散。
如前所述,他們一共嘗試了 LAMP 和 RCA 這兩種等溫擴增方法。由於 LAMP 生成的產物比較複雜,所以他們並未得到較純的目標 DNA 產物。令人興奮的是,RCA 能夠給出清晰的目標產物。於是,他們圍繞 RCA 重新設計了 DNA 顯微鏡技術,即設計出了立體 DNA 顯微鏡技術,並對其進行了系統性優化。
最初的實驗設計如下:
- 第一,使用 poly(T)原位逆轉錄 RNA 生成 cDNA;
- 第二,使用 cDNA 的 3 端連接兩種 RCA 引物;
- 第三,將含有不同 UMI 的兩類單鏈環狀 DNA 退火結合到 RCA 引物;
- 第四,使用滾環擴增方法生成 DNA 納米球,進而編碼 RNA 的空間信息(UMI-cDNA);
- 第五,由於鄰近 DNA 納米球之間橋聯含有 UEI 的 DNA 分子,因此將相鄰 DNA 納米球上的 UMI 拷貝到含 UEI 的 DNA 分子上生成 UMI-UEI-UMI,從而記錄 DNA 納米球的鄰近關係;
- 第六,測序 UMI-cDNA 和 UMI-UEI-UMI,並分析測序數據。
以上便是他們的設計雛形,基本原理是通過 DNA 納米球來標記 RNA 的空間信息,以及通過鄰近 DNA 納米球之間形成橋聯分子來標記其鄰近關係。
圍繞上述設計雛形,他們開始了爲期大約一年的優化過程。依託在培養細胞上的便利性,最初錢年超使用培養細胞來對設計中的每一個酶促反應進行優化。他和合作者一般做到 RCA 產生 DNA 納米球爲止,以便確定設計前半段的反應條件。由於在 RCA 反應中加入了熒光色素標記的 dUTP,因此所產生的 DNA 納米球會帶有熒光。經過 RCA 反應之後,在熒光顯微鏡之下,通過檢測熒光信號的強弱就可以判斷反應條件的好壞。
完成前半段的反應條件優化後,錢年超開始進行端到端的實驗,由於他已經知道 UMI-cDNA 和 UMI-UEI-UMI 這些預期產物的片段大小,因此通過瓊脂糖凝膠電泳來檢測端到端實驗的產物,就可以基本確定反應條件的好壞。然後,通過進一步測序和分析測序數據,就可以定量產物的多少。
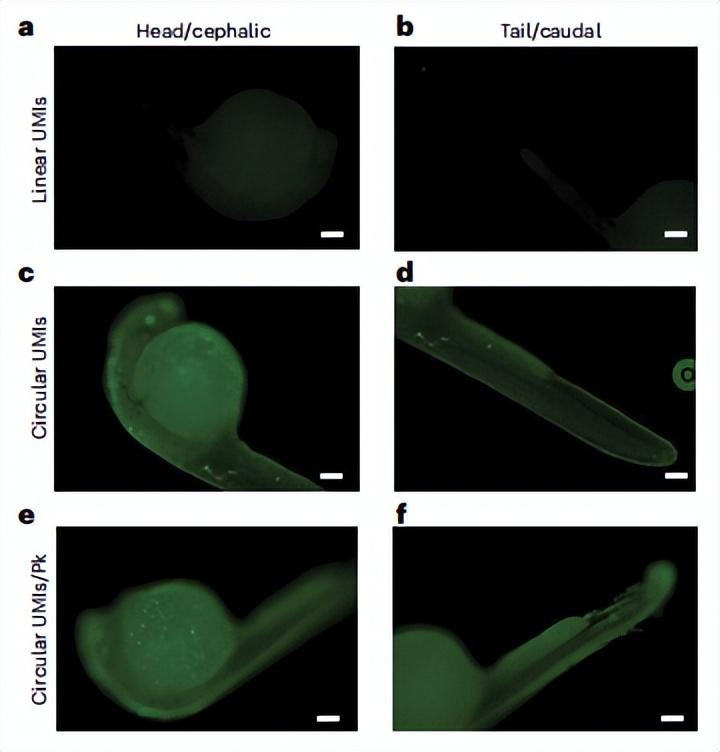
通過使用培養細胞將實驗條件優化之後,接下來錢年超轉向了用受精後 24 小時(24hpf)斑馬魚胚胎做基準測試。因爲斑馬魚胚胎很容易大量獲取,也是研究早期胚胎髮育的有力模型,以及基於培養細胞與三維斑馬魚胚胎在細胞數量、試劑通透性等方面的差異,錢年超用斑馬魚胚胎又進行了爲期一年左右的優化實驗。這時,他終於可以用測序數據重構斑馬魚胚胎的三維形態。
(來源:Nature Biotechnology)
UMI-UEI-UMI 的產量決定了形態重構的成功與否。但是,他得到的 UMI-cDNA 產量依然很低,因爲 24hpf 斑馬魚胚胎有大概 25,000 以上的細胞,每個細胞檢測 400 個不同 UMI-cDNA,就需要 1,000 萬個 UMI-cDNA。爲了解決這一挑戰,錢年超又進一步優化了實驗條件,主要通過引入了“DNA 納米球降解”和“體外轉錄”這兩個核心策略。其表示,DNA 納米球降解可爲後續酶反應釋放納米球所佔的空間,方便酶促反應的組分擴散;體外轉錄可以增加產物的拷貝數,減少丟失產物的風險。
經過精心的實驗設計和多輪的實驗條件優化,他最終成功地開發出了立體 DNA 顯微鏡,目前可以構建 24hpf 斑馬魚胚胎的三維空間轉錄組。

圖 | 相關論文(來源:Nature Biotechnology)
日前,相關論文以《使用立體 DNA 顯微鏡對完整生物進行空間轉錄組成像》(Spatial transcriptomic imaging of an intact organism using volumetric DNA microscopy)爲題發表在 Nature Biotechnology[1],錢年超是第一作者,約書亞·溫斯坦(Joshua A. Weinstein)擔任通訊作者。
參考資料:
1.Qian, N., Weinstein, J.A. Spatial transcriptomic imaging of an intact organism using volumetric DNA microscopy. Nat Biotechnol (2025). https://doi.org/10.1038/s41587-025-02613-z
運營/排版:何晨龍